



RNA测序
介绍相较于一个静态的染色体而言,细胞内的转录物组是一个处于不断变化的动态过程。随着现在的下一代基因测序(NGS)技术的发展,使得可测得的DNA碱基覆盖面增加且样本输出的吞吐量增大。有助于对细胞内RNA转录物进行测序,提供包括选择性剪接的转录、转录后的改变、基因融合、突变/SNPs以及基因表达量改变等细节。,RNA测序不仅能检测mRNA的转录,还能观测到包括包括总RNA和小RNA(miRNA、tRNA和核糖体RNA)在内不同尺度的RNA表达谱。RNA测序还能用来确定外显子/内含子的边界,修正之前注释的5"和3"端基因边界。未来的RNA测序研究还包括观察感染时细胞传导路径的变化和癌症中不同基因表达程度。下一代基因测序之前,对转录物组学和基因表达的研究主要基于基因表达芯片(微阵列),后者包含数以千计用于探测靶向序列的DNA探针,可以得到所有表达出转录物的表达谱。基因表达芯片之后,基因表达的系列分
介绍
相较于一个静态的染色体而言,细胞内的转录物组是一个处于不断变化的动态过程。随着现在的下一代基因测序(NGS)技术的发展,使得可测得的DNA碱基覆盖面增加且样本输出的吞吐量增大。有助于对细胞内RNA转录物进行测序,提供包括选择性剪接的转录、转录后的改变、基因融合、突变/Ss以及基因表达量改变等细节。,RNA测序不仅能检测mRNA的转录,还能观测到包括包括总RNA和小RNA(miRNA、tRNA和核糖体RNA)在内不同尺度的RNA表达谱。RNA测序还能用来确定外显子/内含子的边界,修正之前注释的5"和3"端基因边界。未来的RNA测序研究还包括观察感染时细胞传导路径的变化和癌症中不同基因表达程度。下一代基因测序之前,对转录物组学和基因表达的研究主要基于基因表达芯片(微阵列),后者包含数以千计用于探测靶向序列的DNA探针,可以得到所有表达出转录物的表达谱。基因表达芯片之后,基因表达的系列分析(英语:Serial analysis of gene expression)(SAGE)是主要的基因分析技术。
相对于RNA测序,基因表达芯片(微阵列)测序结果的覆盖面很窄,只能覆盖染色体中1千多万S中的常见等位基因的S(50万到200万)。因此,现有数据库中一般没有罕见等位基因的测序结果,而只有常见的S的数据,这对研究者来说是一个重大缺陷。很多癌症源于突变概率小于1%的突变,因而很难被检测出。但是,基因表达芯片(微阵列)测序在已知的等位基因检测中仍很重要,使它们非常适合监管机构批准的诊断,如囊性纤维化。
免责声明:以上内容版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。感谢每一位辛勤著写的作者,感谢每一位的分享。
——— 没有了 ———
编辑:阿族小谱

文章价值打分
- 有价值
- 一般般
- 没价值
当前文章打 0 分,共有 0 人打分
文章观点支持

0

0
文章很值,打赏犒劳一下作者~

发表评论
写好了,提交
{{item.label}}
{{commentTotal}}条评论
{{item.userName}}
发布时间:{{item.time}}
{{item.content}}
回复
举报


打赏作者
“感谢您的打赏,我会更努力的创作”
— 请选择您要打赏的金额 —
{{item.label}}


{{item.label}}

打赏成功!
“感谢您的打赏,我会更努力的创作”
返回
打赏
私信
推荐阅读
· RNA干扰

发现在转基因的矮牵牛花中所观察到的RNA干扰现象RNA干扰现象是1990年由约根森(Jorgensen)研究小组在研究查尔酮合成酶对花青素合成速度的影响时所发现,为得到颜色更深的矮牵牛花而过量表达查尔酮合成酶,结果意外得到了白色和白紫杂色的矮牵牛花,并且过量表达查尔酮合成酶的矮牵牛花中查尔酮合成酶的浓度比正常矮牵牛花中的浓度低50倍。约根森推测外源转入的编码查尔酮合成酶的基因同时抑制了花中内源查尔酮合成酶基因的表达。1992年,罗马诺(Romano)和Macino也在粗糙链孢霉中发现了外源导入基因可以抑制具有同源序列的内源基因的表达。1995年,Guo和Kemphues在线虫中也发现了RNA干扰现象。1998年,安德鲁·法厄(AndrewZ.Fire)等在秀丽隐杆线虫(C.elegans)中进行反义RNA抑制实验时发现,作为对照加入的双链RNA相比正义或反义RNA显示出了更强的抑制效果。从...
· DNA测序
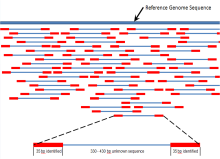
用途历史基本方法Maxam-Gilbert测序法马克萨姆-吉尔伯特测序(英语:Maxam-Gilbertsequencing)是一项由阿伦·马克萨姆(英语:AllanMaxam)与沃尔特·吉尔伯特于1976~1977年间开发的DNA测序方法。此项方法基于:对核碱基特异性地进行局部化学改性,接下来在改性核苷酸毗邻的位点处DNA骨架发生断裂。Sanger测序法Sanger(桑格)双脱氧链终止法是弗雷德里克·桑格(FrederickSanger)于1975年发明的。测序过程需要先做一个聚合酶连锁反应(PCR)。PCR过程中,双脱氧核苷酸可能随机的被加入到正在合成中的DNA片段里。由于双脱氧核糖核苷酸又少了一个氧原子,一旦它被加入到DNA链上,这个DNA链就不能继续增加长度。最终的结果是获得所有可能获得的、不同长度的DNA片段。目前最普遍最先进的方法,是将双脱氧核糖核苷酸进行不同荧光标记。将PCR...
· 桑格测序

原理双脱氧链终止法采用DNA复制原理。Sanger测序反应体系中包括目标DNA片断、脱氧三磷酸核苷酸(dNTP)、双脱氧三磷酸核苷酸(ddNTP)、测序引物及DNA聚合酶等。测序反应的核心就是其使用的ddNTP:由于缺少3"-OH基团,不具有与另一个dNTP连接形成磷酸二酯键的能力,这些ddNTP可用来中止DNA链的延伸。此外,这些ddNTP上连接有放射性同位素或荧光标记基团,因此可以被自动化的仪器或凝胶成像系统所检测到。测序反应过程早期基于Sanger法的测序设置了四个平行的测序反应,每一个反应中分别包括目标片段、DNA聚合酶、测序引物、四种dNTP和一种特定类型萤光(或放射性同位素)标记的ddNTP(即不同的反应中分别为ddATP、ddGTP、ddCTP及ddTTP)。透过这样的配置,在不同的反应中DNA链将会分别在A、G、C及T位中止,并形成不同长度的DNA片断。这些片断随后可被凝胶...
· RNA剪接
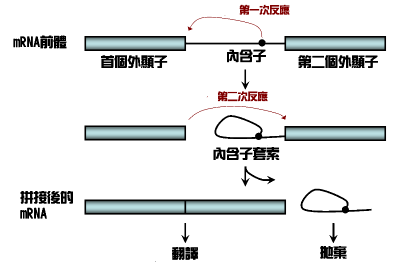
剪接途径RNA剪接可以有多种的方式。剪接的型式以内含子的结构及剪接所需的剪接因子而定。此外,RNA剪接还分为分子内(intramolecular)剪接(cissplicing)以及分子间(intermolecular)剪接(transsplicing)。但不论哪一种途径,移除的内含子都会被抛弃。剪接体内含子经常存在于真核生物的蛋白质编码基因(codinggene)中。在内含子里,需要有5"剪接位点(5"splicesite)、3"剪接位点(3"splicesite)及剪接分枝位点(branchpoint)来进行剪接。剪接是由剪接体(Spliceosome)来催化,它是以五个不同的小核核糖核酸(snRNs)以及不下于一百个蛋白质所组成的大型核糖核酸蛋白质复合物,称为小核核糖蛋白(snRNP)。snRNP的RNA会与内含子行杂交反应(hybridization),并且参与剪接的催化反应。自剪接
· RNA聚合酶
控制转录人类RNA聚合酶I、Ⅱ及Ⅲ的必要亚基控制转录过程会影响基因表达的模式,并从而容许细胞适应不同的环境、执行生物内独特的角色及维持生存所需的代谢过程。所以,RNA聚合酶是活动不单是复杂,而且是有高度规律的。在大肠杆菌中,已确认超过100个因子可以修饰RNA聚合酶的活动。RNA聚合酶可以在特定的DNA序列,称为启动子发动转录。它继而产生RNA链以补足DNA的模板股。并会加入核苷酸至RNA股,这个过程称为“延伸”。在真核生物的RNA聚合酶可以建立一条长达240万个核苷的链(等同于肌萎缩蛋白基因的总长度)。RNA聚合酶会优先在基因末端已编码的DNA序列(称为终结子)释放它的RNA转录本。核糖体会把一些RNA分子会作为蛋白质生物合成的模板。其他会折叠成核酶或转运RNA(tRNA)分子。第三种可能性是RNA分子会单纯地用作控制调节将来的基因表达。(参考小干扰性RNA)RNA聚合酶完成一个全新的合...
关于我们

关注族谱网 微信公众号,每日及时查看相关推荐,订阅互动等。
APP下载

下载族谱APP 微信公众号,每日及时查看

扫一扫添加客服微信















